Professional Documents
Culture Documents
GMX Com
Uploaded by
Himanshu Bisht0 ratings0% found this document useful (0 votes)
35 views1 pageGMX
Original Title
GMX_COM
Copyright
© © All Rights Reserved
Available Formats
TXT, PDF, TXT or read online from Scribd
Share this document
Did you find this document useful?
Is this content inappropriate?
Report this DocumentGMX
Copyright:
© All Rights Reserved
Available Formats
Download as TXT, PDF, TXT or read online from Scribd
0 ratings0% found this document useful (0 votes)
35 views1 pageGMX Com
Uploaded by
Himanshu BishtGMX
Copyright:
© All Rights Reserved
Available Formats
Download as TXT, PDF, TXT or read online from Scribd
You are on page 1of 1
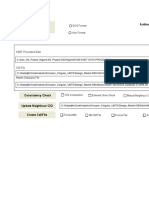
;gmx pdb2gmx -f clean_model.pdb -o model.
gro -water spc
;gmx editconf -f model.gro -o box.gro -d 0.8 -bt cubic
;gmx solvate -cp box.gro -cs spc216.gro -o solv.gro -p topol.top
;gmx grompp -f em.mdp -c solv.gro -p topol.top -o ions.tpr -maxwarn 3
;gmx genion -s ions.tpr -o solv_ions.gro -p topol.top -pname NA -nname CL -neutral
-conc 0.2
;gmx grompp -f em.mdp -c solv_ions.gro -p topol.top -o em.tpr
;gmx mdrun -v -deffnm em
;gmx grompp -f em-cg.mdp -c em.gro -p topol.top -o em-cg.tpr
;gmx mdrun -v -deffnm em-cg -nt 4
;gmx grompp -f nvt.mdp -c em-cg -r em-cg.gro -p topol.top -o nvt.tpr -maxwarn 3
;gmx mdrun -deffnm nvt
;gmx grompp -f npt.mdp -c nvt.gro -r nvt.gro -t nvt.cpt -p topol.top -o npt.tpr -
maxwarn 3
;gmx mdrun -deffnm npt
;gmx grompp -f md.mdp -c npt.gro -t npt.cpt -p topol.top -o md_0.tpr
;gmx mdrun -deffnm md_0
;gmx trjconv -s md_0.tpr -f md_0.xtc -o md_0PBC.xtc -pbc mol -center (1and0)
;gmx rms -s md_0.tpr -f md_0PBC.xtc -o rmsd.xvg -tu ns (4 and 4)
;xmgrace rmsd.xvg
;gmx rmsf -f md_0.xtc -s md_0.tpr -o rmsf.xvg -oq bfac.pdb
;xmgrace rmsf.xvg
;pymol bfac.pdb
;Initital Structure: gmx rms -s em.tpr -f md_0PBC.xtc -o rmsd_xtal.xvg -tu ns
(Choose 4 and 4)
;Structure post MD: gmx rms -s md_0.tpr -f md_0PBC.xtc -o rmsd.xvg -tu ns (Choose 4
and 4)
;Plot the two together using Xmgrace: xmgrace rmsd_xtal.xvg rmsd.xvg
;( where em.tpr is the initial structure and md_0.tpr is after nvt npt and md)
;Black = New Structure; Red =Old Structure
;gmx trjconv -f em.gro -o em.pdb -pbc mol -s em.tpr (gmx to pdb ) (select 1)
; gmx pdb2gmx -f em.pdb -water spce -ignh ( if error occurs)
;gmx energy -s em-cg.tpr -f md_0.edr -o pos.xvg
;xmgrace pos.xvg
obabel inverted_Q1lig1.pdb -O lig2.pdbqt
You might also like
- MD SimDocument3 pagesMD SimGsbsggsbsjysbsnshsbNo ratings yet
- Em Steep CGDocument4 pagesEm Steep CGGsbsggsbsjysbsnshsbNo ratings yet
- RTL Sim CommandsDocument17 pagesRTL Sim CommandsRavi GoyalNo ratings yet
- Dokumen Tipsa26303952Document80 pagesDokumen Tipsa26303952QuocMyNo ratings yet
- 3G Parameters Recomended SettingDocument41 pages3G Parameters Recomended Settingkapoor.nirmalNo ratings yet
- MD Simulation Tutorial in GromacsDocument9 pagesMD Simulation Tutorial in GromacsSimanta PaulNo ratings yet
- Dokumen - Tips Command 1Document101 pagesDokumen - Tips Command 13GSERO100% (1)
- User JVM ArgsDocument1 pageUser JVM ArgsȘTEFAN VALENTIN ANDREINo ratings yet
- Comandos NavigatorDocument20 pagesComandos NavigatorXXXChitaoXXXNo ratings yet
- Optimize Minecraft FPSDocument1 pageOptimize Minecraft FPScesarNo ratings yet
- Find commands to manage radio network elementsDocument10 pagesFind commands to manage radio network elementsSanjay BhelwarNo ratings yet
- Ericsson Command Line Scripts for Cell TroubleshootingDocument39 pagesEricsson Command Line Scripts for Cell TroubleshootingjulescarrelNo ratings yet
- Ejercicio 1 3Document17 pagesEjercicio 1 3SamuelNo ratings yet
- Commands Sheet - NewDocument34 pagesCommands Sheet - Newmausam_panchal2013No ratings yet
- 2g 3g CommandDocument13 pages2g 3g Commandasemaamw workuNo ratings yet
- Command OSS WinfiolDocument9 pagesCommand OSS WinfiolSoemarnaNo ratings yet
- MOLECULAR DYNAMICS Simulations of An ANDROGEN RECEPTOR Using GromacsDocument10 pagesMOLECULAR DYNAMICS Simulations of An ANDROGEN RECEPTOR Using Gromacs@lsreshtyNo ratings yet
- OPTIMIZED PROCEDURESDocument8 pagesOPTIMIZED PROCEDURESdreams125No ratings yet
- All About GSM TechnologyDocument5 pagesAll About GSM Technologysunil_020No ratings yet
- Sts Example CMDDocument2 pagesSts Example CMDjoseph_2707No ratings yet
- New Standard Paremeter 2G 3G 4G 20170417Document28 pagesNew Standard Paremeter 2G 3G 4G 20170417Fahmi YasserNo ratings yet
- Balloon ManualDocument7 pagesBalloon ManualJose GonzalezNo ratings yet
- Confd CrashDocument8 pagesConfd CrashRAMASAMYNo ratings yet
- MSC STSDocument2 pagesMSC STSNitil KumarNo ratings yet
- GROMACS Distance Restraints SetupDocument7 pagesGROMACS Distance Restraints SetupPaula DelgadoNo ratings yet
- MoshellDocument23 pagesMoshellJamie Taylor100% (1)
- PSH CBF350 A4Document1 pagePSH CBF350 A4Handrei MertvykhNo ratings yet
- TechTip - Analyze Your Programs and Applications, Part II - System Administration - IT Infrastructure 3Document6 pagesTechTip - Analyze Your Programs and Applications, Part II - System Administration - IT Infrastructure 3Johnny SaysBlowmeNo ratings yet
- Check and Manage Ericsson BTS CommandsDocument3 pagesCheck and Manage Ericsson BTS CommandsSubodh Mandal0% (1)
- RANCM Param SDR FDD WahyudialiP844023 20230913141059Document2,056 pagesRANCM Param SDR FDD WahyudialiP844023 20230913141059Zulfikar SyariefNo ratings yet
- Command NavigatorDocument20 pagesCommand NavigatorferriyantoNo ratings yet
- Ericsson Bts CommandsDocument4 pagesEricsson Bts CommandsRakesh OinamNo ratings yet
- OSS CommandsDocument7 pagesOSS CommandsHassane AlyNo ratings yet
- Ns2 Streaming Implementations Tutorial VCarrascalDocument11 pagesNs2 Streaming Implementations Tutorial VCarrascalnoormohd99No ratings yet
- LTE MO CommandsDocument31 pagesLTE MO CommandskumarNo ratings yet
- Gromacs Tutorial For Drug Enzyme ComplexDocument21 pagesGromacs Tutorial For Drug Enzyme ComplexManoj Kumar RoutNo ratings yet
- Go program analysisDocument66 pagesGo program analysisSnOw DsignNo ratings yet
- OSS Citrix CommandsDocument2 pagesOSS Citrix CommandsfaysaljiNo ratings yet
- Some Useful Commands in NocDocument9 pagesSome Useful Commands in NocJonan Kansiime100% (1)
- AIX TipsDocument16 pagesAIX TipsClaudio RafaelNo ratings yet
- BSC CommandsDocument1 pageBSC Commandsmaih_wNo ratings yet
- Code For MainDocument3 pagesCode For Mainapi-245348394No ratings yet
- All XRefDocument29 pagesAll XRefrachmat99100% (1)
- RBS COMMANDE CHECKDocument9 pagesRBS COMMANDE CHECKn70No ratings yet
- Command 1Document141 pagesCommand 1Harlen Hutahaean67% (3)
- Oracle SamplesDocument39 pagesOracle Samplesapi-3747607No ratings yet
- MQ CommandsDocument8 pagesMQ Commandssurya_skNo ratings yet
- Sib 2 SsDocument4 pagesSib 2 Ssfar.rhm2021No ratings yet
- GSM OSS Commands: Agonafir Ayele SER So-Em 10.206.115.198 Etagoaye Ethio12345Document2 pagesGSM OSS Commands: Agonafir Ayele SER So-Em 10.206.115.198 Etagoaye Ethio12345Ayele MitkuNo ratings yet
- HUW LTE ParamatersDocument541 pagesHUW LTE ParamatersMuhammad Irfan Hassan Khan0% (1)
- GSM Reconfiguration TG 365 La02824e Air32Document3 pagesGSM Reconfiguration TG 365 La02824e Air32Viren BansalNo ratings yet
- Turbo C Code GpsDocument9 pagesTurbo C Code GpssathyatnNo ratings yet
- Learn Digital and Microprocessor Techniques On Your Smartphone: Portable Learning, Reference and Revision Tools.From EverandLearn Digital and Microprocessor Techniques On Your Smartphone: Portable Learning, Reference and Revision Tools.No ratings yet