Professional Documents
Culture Documents
DavidTutorial 2015
DavidTutorial 2015
Uploaded by
Gana Ibrahim HassanOriginal Description:
Original Title
Copyright
Available Formats
Share this document
Did you find this document useful?
Is this content inappropriate?
Report this DocumentCopyright:
Available Formats
DavidTutorial 2015
DavidTutorial 2015
Uploaded by
Gana Ibrahim HassanCopyright:
Available Formats
Last updated: June, 2015
Tutorial: Using DAVID bioinformatics resources
http://david.abcc.ncifcrf.gov
DAVID bioinformatics resources is an integrated biological knowledgebase and analytical tool
aimed at extracting biological meaning from large gene/protein lists generated from a variety of
high-‐throughput genomic experiments. This tutorial will be conducted starting with a set of 387
probe IDs from an experiment done with peripheral blood mononuclear cells treated with an HIV
envelope protein (gp120) and genome-‐wide expression changes were observed using Affymetrix
U95A microarray chips.
Protocol 1: Uploading a list of gene or probe identifiers
1) Starting DAVID
Open the home page for the DAVID resources site and then click on the link at the top that says
Start Analysis
2) Uploading gene lists into DAVID.
In a second tab, open the Week 2 home page and click on the link for the GenesforGO from the
course website. This is some from a colon cancer study. Either open them and copy and paste or
use the Choose File button to upload the list of genes into the text box below A: Paste a List as
shown in Figure 1.
Select OFFICIAL_GENE_SYMBOL under the drop down menu options
for Select Identifier and select Gene List under the List Type options.
Then click the Submit List button. The Analysis Wizard should say
that multiple species have been detected and and the menu on the
left-‐hand side will switch to the Gene List Manager tab. Highlight
Homo Sapiens (487) and click Select Species. Under the List Manager,
there should be a line that reads genesforGO. You can rename a list
by clicking the Rename button and call it something else.
You can also upload a file using the B: Choose From a File if you have
your identifiers in a text file.
3) Gene name Batch Viewer.
Click on the Shortcut to DAVID tools menu option at the top of the
page and select the menu option Gene Name Batch Viewer.
This should change the page to a Gene List Report as shown in Figure
2 below.
Figure 1: Upload gene list menu
BCH-M628 2015 David Bioinformatics Resources tutorial Page 1 of 5
Last updated: June, 2015
Figure
2:
Gene
List
Report
from
DAVID.
The
gene
names
are
hot
links
to
pages
with
information
about
the
genes.
The
RG
links
will
display,
in
a
new
tab,
a
list
of
other
genes
WITHIN
the
submitted
list
that
are
related
to
the
gene
you
clicked
on.
These
relationships
are
based
on
a
statistical
measure
of
functional
similarity.
Note:
Not
all
genes
will
have
functionally
related
genes
within
the
submitted
list.
Within
the
RG
tab,
you
can
expand
the
scope
of
related
genes
search
under
the
Options
menu.
Change
it
from
User
to
Mus
musculus
and
then
click
the
Rerun
Using
Options
button.
Now
the
search
will
find
any
genes
in
the
mouse
genome
that
are
functionally
related
to
the
one
in
the
list.
3)
Gene
functional
classification.
Return
to
the
tab
with
your
gene
list.
Under
the
Shortcut
to
DAVID
tools menu
option
select
the
option
Gene
Functional
Classification
This
should
open
a
window
displaying
18
clusters
of
gene
groups.
Each
cluster
is
an
attempt
by
DAVID
to
classify
the
submitted
list
of
genes
into
functionally
related
gene
groups.
These
groups
are
identified
by
annotation
term
co-‐occurrence.
The
blue
bars
at
the
top
of
each
cluster
contain
a
number
of
links
that
allow
you
to
learn
more
about
the
clusters
were
identified.
Gene
Group
1
contained
76
genes,
but
just
looking
at
the
gene
names
may
not
tell
you
why
all
of
these
genes
are
grouped
together.
Although
in
this
case,
My
gene
group
1
had
kinesisns,
cyclins
and
kinases.
Click
on
the
red
T
located
in
the
blue
bar
at
the
top
of
the
cluster.
This
will
open
a
new
tab
called
Term
Report.
This
report
lists
all
of
the
terms
annotated
to
these
76
genes
and
the
number
of
genes
annotated
with
a
given
term.
From
this
list
of
terms,
it
seems
most
of
these
genes
are
kinases
or
related
to
kinases.
The
category
column
lists
the
source
provenance
for
the
term.
One
advantage
of
DAVID
to
some
other
gene
ontology
analysis
programs
is
that
it
uses
annotation
information
from
a
wider
variety
of
sources.
Clicking
on
the
blue
bar
in
the
column
titled
Genes
in
Group
will
open
a
tab
with
a
list
of
the
genes
annotate
with
a
given
term.
Return
to
the
tab
Gene
Functional
Classification
results
tab.
You
may
want
to
close
some
of
the
tabs
that
DAVID
opened,
as
the
browser
can
get
fairly
cluttered.
4)
Functional
Annotation
Chart
BCH-M628 2015 David Bioinformatics Resources tutorial Page 2 of 5
Last updated: June, 2015
Under
the
Shortcut
to
DAVID
tools menu
option
select
the
option
Functional
annotation.
This
will
open
a
window
titled
Annotation
Summary
Results
and
has
a
lot
of
different
options.
This
is
the
main
window
by
which
you
can
do
an
enrichment
analysis
with
DAVID.
Use
the
default
options
and
click
the
Functional
Annotation
Chart
button
located
at
the
bottom
of
the
page.
This
will
open
a
new
window
(rather
than
a
new
tab)
as
shown
in
Figure
3
below.
Figure
3:
Functional
annotation
chart
In
the
functional
annotation
chart,
each
annotation
term
associated
with
the
list
of
genes
is
tested
for
enrichment
relative
to
the
background
set
of
genes.
In
this
case
there
are
1200
chart
records.
The
chart
lists
the
terms
that
are
statistically
enriched,
in
order
of
increasing
Benajmini
adjusted
p-‐value,
located
in
right-‐most
column.
You
can
adjust
which
columns
of
data
are
shown
under
the
Options
menu
located
above
the
list.
The
entire
file
can
be
downloaded
in
a
format
compatible
with
Excel.
The
Count
and
%
columns
represent
the
number
of
genes
in
the
list
annotated
with
the
term
and
percent
of
genes
in
the
list
annotated
with
that
term.
So
for
the
SP_PIR_KEYWORD
term
“phosphoprotein”
there
are
316
genes
in
the
list
of
487,
or
65%
of
the
total
list.
If
you
want
to
see
what
genes
are
annotate
with
that
term,
click
on
the
purple
bar
under
the
Genes
column
and
it
will
open
a
tab
with
a
list
of
all
the
genes
annotated
with
that
term
in
the
list.
This
type
of
term
enrichment
is
very
similar
to
the
results
you
would
get
from
other
GO
analysis
programs,
with
the
added
benefit
of
additional
annotation
sources.
One
thing
you
have
probably
already
noticed
is
that
the
different
terms
have
a
fair
amount
of
overlap
with
regard
to
their
function
or
role
in
the
cell.
For
example
two
INTERPRO
terms
are
“protein
kinase,
core”
with
67
genes
and
“Protein
kinase,
ATP
binding
site”
with
65
genes.
It’s
likely
that
most
of
the
6
genes
in
these
two
terms
overlap.
This
is
a
frustrating
aspect
of
conducting
a
gene
ontology
analysis.
That
is
knowing
when
and
how
to
collapse
terms
to
remove
some
of
the
redundancy.
DAVID
has
attempted
to
address
this
issue
with
another
tool
called
Functional
Annotation
Clustering.
5)
Functional
Annotation
Clustering
Return
to
the
window
and
tab
Functional
Annotation
Tools
and
click
on
the
Functional
Annotation
Clustering
button
located
at
the
bottom
of
the
page.
This
will
open
another
new
window
as
shown
in
Figure
4.
I
got
240
clusters
when
I
did
this
analysis.
BCH-M628 2015 David Bioinformatics Resources tutorial Page 3 of 5
Last updated: June, 2015
Figure 4: Functional annotation clustering window
Now
instead
of
a
long
list
of
single
terms,
you
see
clusters
of
terms
that
are
related
to
each
other.
The
clusters
are
shown
in
order
of
decreasing
enrichment
scores.
If
you
click
on
the
red
G
button
located
on
the
blue
bar
at
the
top
of
each
cluster,
you
should
get
a
list
of
the
genes
in
that
cluster
that
are
annotated
with
one
or
more
of
the
terms.
NOTE:
the
number
of
genes
is
not
going
to
be
a
sum
of
the
Count
for
each
term
because
many
of
the
genes
will
be
annotated
with
more
than
one
term
listed
in
the
cluster.
You
can
alter
the
options
and
stringency
of
the
cluster
generation
and
rerun
the
analysis.
Increasing
the
stringency
will
usually
decrease
both
the
number
of
clusters
and
the
number
of
genes
in
the
clusters.
In
this
case,
when
I
changed
the
stringency
from
medium
to
high,
it
returned
191
clusters
but
with
many
few
terms
per
cluster.
The
first
cluster
went
from
170
to
111
genes.
This
is
where
the
analysis
becomes
much
more
involved.
There
is
NOT
going
to
be
one
answer
or
even
one
approach
to
this
type
of
analysis.
This
type
of
analysis
provides
a
guide
to
new
ways
of
thinking
about
the
data,
but
is
not
going
to
pinpoint
the
2-‐5
genes
that
will
form
the
core
of
a
paper.
That’s
up
to
you
and
your
ability
to
synthesize
information
from
this
as
well
as
other
sources.
That
is,
here
is
where
you
need
to
really
think
about
your
data
and
what
it
means
within
the
context
of
your
experimental
model
and
in
the
larger
context
of
other
published
data.
6)
Creating
a
sublist
Suppose
you
find
a
functional
cluster
that
is
of
particular
interest.
You
may
want
to
follow
up
on
the
genes
in
that
cluster
in
more
depth.
To
do
that,
check
the
boxes
located
to
the
left
of
each
term
that
you
want
to
include
in
the
sublist.
Then
click
the
Create
Sublist
button.
It
will
give
you
an
option
to
give
it
a
more
descriptive
name,
then
click
OK.
After
that,
the
new
list
should
show
up
in
the
List
Manager.
You
can
select
it
and
click
the
Use
button
to
do
a
more
in-‐depth
analysis
on
the
new
list.
7)
GeneID
conversion
In
case
you
didn’t
get
enough
of
this
during
the
BioMart
tutorial,
here’s
another
opportunity.
Select
the
original
GenesforGO
under
List
Manager.
Under
the
Shortcut
to
DAVID
tools,
choose
the
Gene
ID
Conversion
tool
option.
For
option
1,
select
the
conversion
output
to
BCH-M628 2015 David Bioinformatics Resources tutorial Page 4 of 5
Last updated: June, 2015
ENTREZ_GENE_ID
then
click
the
Submit
to
Conversion
Tool
button.
Out
of
487,
all
486
were
successfully
converted.
The
output
should
give
you
a
ice
list
of
the
Gene
symbols
(in
the
From
column)
and
Entrez
GeneID
numbers
(To
column)
and
the
gene
name.
The
option
exists
to
download
this
file
in
a
format
compatible
with
Excel.
8)
General
annotation
information
You
can
also
use
the
Functional
Annotation
tools
to
obtain
more
general
information
about
each
of
your
genes.
Return
to
the
Functional
Annotation
Tool
tab.
Expand
the
menu
under
General
Annotations.
Click
on
the
blue
bar
to
the
right
of
ENTREZ_GENE_SUMMARY.
You
should
see
a
new
window
pop
up
with
data
for
356
of
the
genes.
There
is
a
summary
paragraph
for
each
probe
ID
that
was
matched
in
the
Entrez
Gene
database
that
also
contained
summary
data.
This
is
probably
more
useful
for
a
smaller
set
of
genes,
but
is
a
quick
way
to
assemble
some
basic
information
about
the
genes
in
whatever
list
you
are
working
with.
DAVID
also
has
links
to
Pathways
and
Protein-‐Protein
interaction
databases
but
we’ll
work
on
those
later
this
week
and
next.
9)
Saving
Charts
from
DAVID
&
importing
into
Excel.
After
you’ve
done
a
functional
annotation
cluster
or
generated
a
chart
for
gene
ontology,
the
data
returned
can
be
downloaded
in
a
tab-‐delimited
text
file.
It
can
be
easily
imported
into
excel.
Click
on
the
Download
File
link
at
the
top
right
of
the
page.
It
will
open
a
new
page
or
tab
with
the
data
in
text
format.
Click
on
the
page
and
then
under
the
browser
File
menu,
select
Save
Page
As…
and
save
it
as
a
text
file
to
your
hard
drive.
Open
Excel,
then
open
the
text
file
from
within
Excel.
It
should
import
nicely,
with
the
data
distributing
itself
into
columns.
There
is
one
caveat
with
the
gene
ontology
data.
It
is
presented
in
DAVID
as
“GOtermID~GOname”
and
when
you
import
it
into
Excel,
that
comes
in
as
a
single
entity.
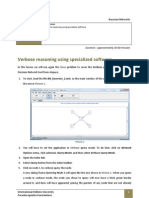
To
split
the
GOtermID
and
GOname
into
two
adjacent
cells,
when
you
get
to
Step
2
of
the
Text
Import
Wizard,
add
the
tilde
(~)
character
as
another
delimiter.
That
is,
check
the
Other
box
under
Delimiters
and
type
a
~
in
the
box
as
shown
below:
This
will
split
your
GOterm
ID
from
the
GOterm
name.
You
will
need
to
adjust
the
column
headings
as
once
you’ve
split
the
GO
data,
the
columns
headings
are
off
by
one.
BCH-M628 2015 David Bioinformatics Resources tutorial Page 5 of 5
You might also like
- Excel Formulas and FunctionsDocument50 pagesExcel Formulas and FunctionsBoypeace Pamotan100% (3)
- Cerner QuickReferenceGuide.2006Document3 pagesCerner QuickReferenceGuide.2006yostyNo ratings yet
- TOS & Test QuestionsDocument51 pagesTOS & Test Questionsagnes100% (2)
- Uco Bank Form Iepf 1Document437 pagesUco Bank Form Iepf 1sandip_exlNo ratings yet
- SC EX 2 Klapore ShilohKnowles Report 2Document3 pagesSC EX 2 Klapore ShilohKnowles Report 2Shiloh KnowlesNo ratings yet
- SPSS HandoutDocument43 pagesSPSS HandoutÖrdinärySoel100% (2)
- Trial Balance (Excel Format)Document4 pagesTrial Balance (Excel Format)Mohammed Shabil50% (2)
- DeltaV v12.3.1 Software Updates PDFDocument53 pagesDeltaV v12.3.1 Software Updates PDFevbaru100% (1)
- Vulnerability Assessment Tool BRC v2.0Document31 pagesVulnerability Assessment Tool BRC v2.0Juan FelipeNo ratings yet
- X'Pert HighScore Plus IntroductionDocument15 pagesX'Pert HighScore Plus IntroductionLevent Kartal100% (1)
- JAMOVI 2017 Statistics For Psychologists Section - JAMOVI Chapter - Using The Software - 1Document25 pagesJAMOVI 2017 Statistics For Psychologists Section - JAMOVI Chapter - Using The Software - 1Carlos EscalhaoNo ratings yet
- Data VisualizationDocument24 pagesData VisualizationVarun GuptaNo ratings yet
- FAO Guide to Ranking Food Safety Risks at the National LevelFrom EverandFAO Guide to Ranking Food Safety Risks at the National LevelNo ratings yet
- MassLynx GuideDocument604 pagesMassLynx GuideDiego García Ríos100% (4)
- Rast TutorialDocument10 pagesRast TutorialPeter JustinNo ratings yet
- Exercise 7 BioinformaticsDocument8 pagesExercise 7 BioinformaticsHiroshi MatsushimaNo ratings yet
- Computer Lab 1 Phylogenetics BSID 2022Document10 pagesComputer Lab 1 Phylogenetics BSID 2022Kiran DuwadiNo ratings yet
- Dchip ExpressionDocument4 pagesDchip ExpressionZoe Ling Ean CheungNo ratings yet
- CMB Lab Exp 9Document9 pagesCMB Lab Exp 9Elizabeth Jade ViceraNo ratings yet
- DAVID TutorialDocument34 pagesDAVID TutorialData ScrewNo ratings yet
- Userguide FAO Ration Tool-EnglishDocument8 pagesUserguide FAO Ration Tool-EnglishIcky IckyNo ratings yet
- Metrologic Instruments, Inc. Metroset: 2, Extended Parsing FeatureDocument12 pagesMetrologic Instruments, Inc. Metroset: 2, Extended Parsing FeatureferNo ratings yet
- Metabolic Reconstruction by RAST ServerDocument8 pagesMetabolic Reconstruction by RAST ServerDian Pribadi Perkasa IbrahimNo ratings yet
- Try It Free BRC v2.0Document31 pagesTry It Free BRC v2.0Brian Rolando Cerros FonsecaNo ratings yet
- Vulnerability Assessment Tool: Food Fraud Advisors 2017Document48 pagesVulnerability Assessment Tool: Food Fraud Advisors 2017Fitriyanti IzinilahNo ratings yet
- Maico Database ManualDocument11 pagesMaico Database ManualJuan Carlos Enríquez GómezNo ratings yet
- PIPE2 TutorialDocument11 pagesPIPE2 TutorialWolelaw EndalewNo ratings yet
- Exploring The Structure and Contents of A Data SetDocument14 pagesExploring The Structure and Contents of A Data SetGokhan OcakogluNo ratings yet
- Try It Free v3.0sDocument49 pagesTry It Free v3.0sj KiizaNo ratings yet
- Typing Epidemiological ClusteringDocument20 pagesTyping Epidemiological ClusteringVahid MansouriNo ratings yet
- Useful ToolsDocument11 pagesUseful ToolsCoccolNo ratings yet
- PARE Introduction To Bioinformatics.v3Document6 pagesPARE Introduction To Bioinformatics.v3Evan LiouNo ratings yet
- Species Delimitation Plugin ManualDocument6 pagesSpecies Delimitation Plugin ManualRicardo GarcíaNo ratings yet
- FDA Vulnerability Assessment Tool Quick Instruction GuideDocument6 pagesFDA Vulnerability Assessment Tool Quick Instruction Guideasanchezt100% (1)
- PubMed Database Interface ExercisesDocument6 pagesPubMed Database Interface ExercisesChhaibi AbderrahimNo ratings yet
- Quick Reference Guide For Search and Patches & Updates: Powerview Product Based SearchDocument2 pagesQuick Reference Guide For Search and Patches & Updates: Powerview Product Based SearchJAy RastaNo ratings yet
- ParaView Getting Started GuideDocument2 pagesParaView Getting Started GuideAldo Dwi RizaldiNo ratings yet
- Xcel ATA AB: Groups/Buttons DescriptionDocument3 pagesXcel ATA AB: Groups/Buttons DescriptionSaing ChochNo ratings yet
- Bayesian Networks Exercise 4Document7 pagesBayesian Networks Exercise 4Konstantinos ParaskevopoulosNo ratings yet
- Getting Started With Cognos 11Document21 pagesGetting Started With Cognos 11Jose Luis Mendoza LandaverryNo ratings yet
- Taxonomic ProfilingDocument13 pagesTaxonomic ProfilingVahid MansouriNo ratings yet
- Setting COVID Batch Numbers For SessionsDocument3 pagesSetting COVID Batch Numbers For SessionsAsim AzizNo ratings yet
- FeedAccess-User Guidebook Formulation PDFDocument17 pagesFeedAccess-User Guidebook Formulation PDFTấn Khoa NguyễnNo ratings yet
- Argus Molecular Docking 1Document5 pagesArgus Molecular Docking 1escherichioNo ratings yet
- User Manual of Webgestalt 2019: January 24, 2019Document33 pagesUser Manual of Webgestalt 2019: January 24, 2019dinaNo ratings yet
- GuidelineDocument7 pagesGuidelineDominic EmerencianaNo ratings yet
- Administrator Guide FAO Ration Tool-EnglishDocument15 pagesAdministrator Guide FAO Ration Tool-EnglishIcky IckyNo ratings yet
- WFOOD2Document231 pagesWFOOD2FirsandiPrasastyaFikryGozaliNo ratings yet
- WFOOD2Document231 pagesWFOOD2FirsandiPrasastyaFikryGozaliNo ratings yet
- WFOOD2Document231 pagesWFOOD2FirsandiPrasastyaFikryGozaliNo ratings yet
- M517-E123 (DAR-7500 DR Console DICOM MPPS)Document12 pagesM517-E123 (DAR-7500 DR Console DICOM MPPS)김진오No ratings yet
- Chapter VDocument21 pagesChapter VRm OtNo ratings yet
- Web Listing of WHO Prequalified VaccinesDocument9 pagesWeb Listing of WHO Prequalified VaccinesArif SetiabudiNo ratings yet
- SPSS Basics ManualDocument25 pagesSPSS Basics ManualAlfredBakChoiNo ratings yet
- Coursera BioinfoMethods-II Lab02Document14 pagesCoursera BioinfoMethods-II Lab02Alex ChenNo ratings yet
- Analyze Viral Hybrid Data and Identify IntegrationDocument12 pagesAnalyze Viral Hybrid Data and Identify IntegrationVahid MansouriNo ratings yet
- Protocols For BioEditDocument24 pagesProtocols For BioEditHadi SastranegaraNo ratings yet
- Task 2 - Biodiversity - Evolution - Genetic VariationsDocument7 pagesTask 2 - Biodiversity - Evolution - Genetic VariationsMuhamad Yazid Bin Siratur RahmanNo ratings yet
- User Manual On Referral Module Revised 280521Document11 pagesUser Manual On Referral Module Revised 280521SrinuNo ratings yet
- Vegetation Analysis ToolkitDocument8 pagesVegetation Analysis ToolkitAshoka VanjareNo ratings yet
- Week 8Document18 pagesWeek 8Master PlanerNo ratings yet
- Bi 0107Document13 pagesBi 0107guiervicalNo ratings yet
- Biochemical Systematic by Using Dna Fingerprint DataDocument17 pagesBiochemical Systematic by Using Dna Fingerprint Datapratiwi kusumaNo ratings yet
- Agilent 1100MSD GettingStartedDocument32 pagesAgilent 1100MSD GettingStartedMarine JolieNo ratings yet
- Discovery Studio VisualizerDocument31 pagesDiscovery Studio VisualizerbitMorph3rNo ratings yet
- Analyzing the Large Number of Variables in Biomedical and Satellite ImageryFrom EverandAnalyzing the Large Number of Variables in Biomedical and Satellite ImageryNo ratings yet
- Integration and Visualization of Gene Selection and Gene Regulatory Networks for Cancer GenomeFrom EverandIntegration and Visualization of Gene Selection and Gene Regulatory Networks for Cancer GenomeNo ratings yet
- Tutorial 03 Storage TankDocument18 pagesTutorial 03 Storage TankObaid Ur RehmanNo ratings yet
- PricingDocument5 pagesPricingAnwesha Dev ChaudharyNo ratings yet
- Active Reports 9Document1,100 pagesActive Reports 9faraonchisNo ratings yet
- MS Access - AssignmentDocument5 pagesMS Access - Assignmentpratham puriNo ratings yet
- Notice: I Have: Engineering CalculationDocument34 pagesNotice: I Have: Engineering CalculationAlexandre GelsiNo ratings yet
- Import DataDocument41 pagesImport DataSaran RavikumarNo ratings yet
- Res Load 11Document18 pagesRes Load 11nguyentienphatNo ratings yet
- EZ Professional CoursesDocument9 pagesEZ Professional CoursesKG LikesNo ratings yet
- EUROCODE 1991-1 Worked ExamplesDocument185 pagesEUROCODE 1991-1 Worked ExamplesJovito ConfesorNo ratings yet
- 251 RpaDocument24 pages251 Rpaxyxy26799No ratings yet
- Data Visualisation For Reporting: Using Microsoft Excel DashboardDocument2 pagesData Visualisation For Reporting: Using Microsoft Excel DashboardJames WarrenNo ratings yet
- Introduction To Excel VBA Programming - SARA EDocument18 pagesIntroduction To Excel VBA Programming - SARA EMike PyanxNo ratings yet
- Uspv 60-01-44-00-00Document28 pagesUspv 60-01-44-00-00imkzbyNo ratings yet
- Histogram in Excel - Easy Excel TutorialDocument5 pagesHistogram in Excel - Easy Excel TutorialJamalodeen MohammadNo ratings yet
- It Word ProjectDocument54 pagesIt Word ProjectTushar BasiaNo ratings yet
- EXCEL SHORT KEYS Pankaj BahukhandiDocument2 pagesEXCEL SHORT KEYS Pankaj BahukhandiPankaj KumarNo ratings yet
- Assignment 1 & 2Document3 pagesAssignment 1 & 2Rajalakshmi MuthuvelNo ratings yet
- Manuale Suite enDocument136 pagesManuale Suite enAntonio GuillenNo ratings yet
- Core Chapter 09 Excel Master 4th Edition StudentDocument77 pagesCore Chapter 09 Excel Master 4th Edition StudentShaunak ChitnisNo ratings yet
- Start and Stop The Microsoft Access - Create New A Database - Database Objects - Design The Table - Data Types - Field PropertiesDocument12 pagesStart and Stop The Microsoft Access - Create New A Database - Database Objects - Design The Table - Data Types - Field PropertiesLoïc JEAN-CHARLESNo ratings yet
- CPF Lab 003 PDFDocument10 pagesCPF Lab 003 PDFMuhammad MuzammilNo ratings yet
- BNS-3513 Research Methodology-Lab Manual-Section 1Document13 pagesBNS-3513 Research Methodology-Lab Manual-Section 1arunkorathNo ratings yet
- Guardian Newsletter January 2015 PDFDocument11 pagesGuardian Newsletter January 2015 PDFpalotito_eNo ratings yet